Rでの生存時間解析(時間変化共変量の処理)
今回は生存時間解析について。
フォローアップ中に、喫煙から非喫煙になったり、運動習慣が変化したり、などとアウトカムに関係のある変数が変化した時に、どう対応すればよいのでしょうか。このように、変化しうる共変量をTime varying covariates / time dependent covariates (時間依存共変量?時間変化共変量?)と言います。
じつは、Survival Analysisにとっては、この時間依存性の共変量の処理は大きく変わったことをしているわけではありません。そのあたりを示せればと思います。 まず、下記のRコードを走らせます。
# Settings
library(survival)
library(rms)
# Data generation
set.seed(1)
test = data.frame(id=1:400,
start=sample(1:100, 400, replace = T),
stop =sample(105:400, 400, replace = T),
outcome=c(rbinom(200, 1, prob=0.1), rbinom(200, 1, prob=0.2)),
treat = c(rep(0, 200), rep(1, 200)),
smoke = c(sample(0:1, 400, replace = T))) # smoke indicator
# Data processing
test$ptime = test$stop - test$start # person time
test$SurvObj1 = with(test, Surv(ptime, outcome == 1)) # using person time
test$SurvObj2 = with(test, Surv(start, stop, outcome == 1)) # using start-stop
test$SurvObj3 = with(test, Surv(rep(0,400), ptime, outcome == 1)) # using 0-ptime
head(test)
# KM curve and ploting
model1 = survfit(SurvObj1 ~ treat, data = test)
## for survplot (survplot has more options)
## model1.1 <- npsurv(SurvObj ~ trea+smoke, data = test, conf.type = "log-log") ;survplot(model1.1)
model2 = survfit(SurvObj2 ~ treat, data = test)
model3 = survfit(SurvObj3 ~ treat, data = test)
par(mfcol=c(1,3))
plot(model1)
plot(model2)
plot(model3)
# Cox proportional hazarmodel
coxph(SurvObj1 ~ treat + smoke, data=test)
coxph(SurvObj2 ~ treat + smoke, data=test)
coxph(SurvObj3 ~ treat + smoke, data=test)
下記の条件でSurvobjに対して、KM曲線を書きます。
SurvObj1はsurvival特有のデータで、ここでは単純にstartとstopの差(人時間)を取っています。(model1)
SurvObj2は人時間自体は同じですが、start timeとstoptimeを残しています。(model2)
- SurvObj3はstart timeをすべて0にして、stoptimeは0+人時間にしました。(model3)
このKMモデルでわかるように、model1(左)とmodel2(中央)は別物です。一方でmodel1とmodel3(右)のグラフは同じであることがわかります。
 生存解析では、人時間(観察期間)だけでなく、どこからStartしてどこでStopしたかという、該当期間の位置も考慮すべきことがわかります。Stopが105から始まっているmodel2の回帰ではtime = [0-104]までイベントが起きていません。
model1 == model3 != model2ということは、コード中のコックス比例ハザードモデルの結果でもわかります(走らせてみてください)。
生存解析では、人時間(観察期間)だけでなく、どこからStartしてどこでStopしたかという、該当期間の位置も考慮すべきことがわかります。Stopが105から始まっているmodel2の回帰ではtime = [0-104]までイベントが起きていません。
model1 == model3 != model2ということは、コード中のコックス比例ハザードモデルの結果でもわかります(走らせてみてください)。
時間依存共変量
さて、start とstopという情報が生存解析に重要であることが見えてきたので、ちょっと面倒ですが、今度は、time-varying covariateを含むデータを作って回帰してみます。
# time varying covariate graph
library(dplyr)
set.seed(113)
test=data.frame(id = 1:400,
baseline= 0,
smoke = c(rbinom(400, 1, prob=0.3)),
treat = c(rep(0, 200), rep(1, 200)),
censor = sample(10:180, 400, replace = T),
outcome = c(rbinom(200, 1, prob=0.1), rbinom(200, 1, prob=0.2)))
test1 = test %>%
mutate(visit1 = sample(21:42, 400, replace = T),
smoke1 = c(rbinom(400, 1, prob=0.3))) %>%
mutate(visit2 = visit1 + sample(21:42, 400, replace = T),
smoke2 = c(rbinom(400, 1, prob=0.3))) %>%
mutate(visit3 = visit2 + sample(21:42, 400, replace = T),
smoke3 = c(rbinom(400, 1, prob=0.3))) %>%
mutate(visit1 = ifelse(visit1<censor, visit1, NA),
smoke1 = ifelse(visit1<censor, smoke1, NA),
visit2 = ifelse(visit2<censor, visit2, NA),
smoke2 = ifelse(visit2<censor, smoke2, NA),
visit3 = ifelse(visit2<censor, visit3, NA),
smoke3 = ifelse(visit2<censor, smoke3, NA))
test2 = tmerge(data1 = test, data2=test1, id=id, tstop=censor, event = event(censor, outcome))
test2 = tmerge(data1 = test2, data2=test1, id=id, smoke=tdc(visit1, smoke1))
test2 = tmerge(data1 = test2, data2=test1, id=id, smoke=tdc(visit2, smoke2))
test2 = tmerge(data1 = test2, data2=test1, id=id, smoke=tdc(visit3, smoke3))
# KM plot
## smoking status fixed at baaseline
test$SurvObj1 = with(test , Surv(censor, outcome == 1))
## smoking status varying time
test2$SurvObj2 = with(test2, Surv(tstart, tstop, event == 1))
par(mfcol=c(1,2))
plot(survfit(SurvObj1~smoke, test))
plot(survfit(SurvObj2~smoke, test2))
# Cox hazard model
coxph(SurvObj1 ~ treat + smoke, data=test)
coxph(SurvObj2 ~ treat + smoke, data=test2)
testデータセットではベースラインデータと、打ち切り(センサー)データ、アウトカムを作り、test1では各visitにおける、喫煙の有無の情報を加えました。
さて、test1は1人1行ですが、これを、各visitごとに別々の観察行にしてやります。この操作にはSurvivalパッケージのtmergeを使います。
time2をじっくり見ていただくと、ひとりの人のビジット毎に観察が分けられ、smokeの状況が更新されていること、イベントが起きた場合にのみevent==1となっていることに気づきます。
下記グラフは、喫煙の有無で、層別化したKM曲線で、左がベースラインの情報のみを使ったもの、右が喫煙状態が変化しているのを考慮したものです*1。
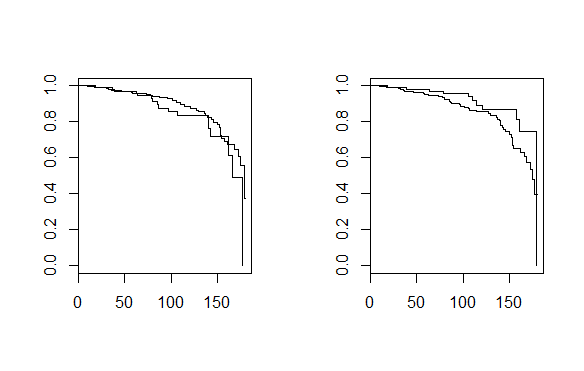
共変量の時間変化を考慮することでグラフの形がかなり変わっています。コックス比例ハザードモデルのパラメータも違うことが上のコードを走らせると確認できます。ただ観察が細切れになっているだけで解析モデルの記述自体は時間共変量を考慮しても考慮しなくても同じであることが分かると思います。
まとめ
時間依存性共変量がある解析は、観察のstartとstopの情報を保持して、一人の人をいくつものオブザーベーションに分割して、その中での共変量の状態を使って回帰していきます。
それ以外のところは通常の回帰と同じです。同じ人が何度もデータに出てくるから独立性は大丈夫なのか!という感じが最初はします。しかし、コックス比例モデルは、ある瞬間のハザード(イベント発生率)に回帰していて、この場合、その瞬間においては症例の重複はないので、問題ないと理解しています。
オブザーベーションを切り分けていくのは自分でやるとかなり面倒ですが、tmergeがあって助かりました。Survivalパッケージのヘルプに、いろいろとtmergeのオプションが紹介されています。
*追記: tmergeの挙動が変わっていて上のコードで上手くtest2ができなくなっています。ちょっとブサイクですが、上記でtestを作ったあとは、下記のようにtest4を作って、使えば動くと思います。
test1 = test %>%
mutate(smoke0=smoke) %>%
mutate(visit1 = sample(21:42, 400, replace = T),
smoke1 = c(rbinom(400, 1, prob=0.3))) %>%
mutate(visit2 = visit1 + sample(21:42, 400, replace = T),
smoke2 = c(rbinom(400, 1, prob=0.3))) %>%
mutate(visit3 = visit2 + sample(21:42, 400, replace = T),
smoke3 = c(rbinom(400, 1, prob=0.3))) %>%
mutate(visit1 = ifelse(visit1<censor, visit1, NA),
smoke1 = ifelse(visit1<censor, smoke1, NA),
visit2 = ifelse(visit2<censor, visit2, NA),
smoke2 = ifelse(visit2<censor, smoke2, NA),
visit3 = ifelse(visit2<censor, visit3, NA),
smoke3 = ifelse(visit2<censor, smoke3, NA))
test2 = tmerge(data1 = test, data2=test1, id=id, tstop=censor, event = event(censor, outcome))
test2 = tmerge(data1 = test2, data2=test1, id=id, smoke=tdc(visit1, smoke1))
test2 = tmerge(data1 = test2, data2=test1, id=id, smoke=tdc(visit2, smoke2))
test2 = tmerge(data1 = test2, data2=test1, id=id, smoke=tdc(visit3, smoke3))
test3 = left_join(test2, test1[c('id', 'baseline', 'smoke0')],
by=c('id', 'tstart'='baseline')) %>%
mutate(smoke=ifelse(!is.na(smoke0), smoke0, smoke))
test3 = left_join(test3, test1[c('id', 'visit1', 'smoke1')],
by=c('id', 'tstart'='visit1')) %>%
mutate(smoke=ifelse(!is.na(smoke1), smoke1, smoke))
test3 = left_join(test3, test1[c('id', 'visit2', 'smoke2')],
by=c('id', 'tstart'='visit2')) %>%
mutate(smoke=ifelse(!is.na(smoke2), smoke2, smoke))
test3 = left_join(test3, test1[c('id', 'visit3', 'smoke3')],
by=c('id', 'tstart'='visit3')) %>%
mutate(smoke=ifelse(!is.na(smoke3), smoke3, smoke))
test4 = test3 %>% select(id, treat, tstart, tstop, smoke, event)
# KM plot
## smoking status fixed at baaseline
test$SurvObj1 = with(test , Surv(censor, outcome == 1))
head(test)
## smoking status varying time
test4$SurvObj2 = with(test4, Surv(tstart, tstop, event == 1))
head(test4)
par(mfcol=c(1,2))
plot(survfit(SurvObj1~smoke, test))
plot(survfit(SurvObj2~smoke, test4))
# Cox hazard model
coxph(SurvObj1 ~ treat + smoke, data=test)
coxph(SurvObj2 ~ treat + smoke, data=test4)
*1:time-lagは考慮していません